蛋白质结构预测(Protein Structure Prediction)
ROSIE (https://rosie.rosettacommons.org/)为Rosetta的Web端,提供许多分子建模与蛋白对接相关的在线软件,包括RNA预测、肽段设计、蛋白-配体对接等多种软件。
1.1 蛋白质的二级结构预测
蛋白质二级结构主要指多肽链中规则的重复构象,常见的二级结构有α螺旋(helix)、β折叠(sheet)、β转角(turn)和无规卷曲(random coil)等。多肽链中氨基酸的非侧链原子构成了蛋白质骨架(backbone)。骨架中共价键的键长和平面键角一般都是固定的,即肽键是刚性的并且处于同一个平面上(肽平面)。在蛋白质骨架中,α碳原子(与侧链相连的C原子)和其它骨架原子之间形成的键里面只有两个是可旋转的(图 8 3):(1)α碳原子与氨基氮原子之间形成的键的旋转角表示为Φ(phi),(2)α碳原子与羰基碳原子之间形成的键的旋转角表示为Ψ(psi)。一个蛋白质的骨架构象可以通过每个氨基酸的Φ与Ψ角来表示。由于一些Φ与Ψ角的组合可能导致骨架上相邻残基侧链原子的空间碰撞,因此物理上并不能观察到所有的Φ与Ψ角。拉氏图(Ramachandran plot)能显示氨基酸残基中允许出现的二面角Φ与Ψ值。

图 8 3 蛋白质骨架 二级结构是指蛋白质分子中多肽链骨架中原子的局部空间结构,并不涉及氨基酸残基侧链的构象。常见的α螺旋的Φ角与Ψ角大约是-60°(Φ≈Ψ≈-60°),呈弹簧状螺旋结构,每一圈有3.6个氨基酸。β折叠具有完全伸展的骨架,Φ角与Ψ角分别是-135°与135°(Φ≈-135°,Ψ≈135°)。β折叠可以和其它β折叠连在一起形成平行β折叠片或反平行β折叠片。β转角是反平行β折叠片中当一个β折叠倒转方向时形成的U形的转角结构。预测一个多肽链序列的三维结构的第一步就是决定骨架的哪些区域可能会形成螺旋,哪些区域会形成折叠,又有哪些区域形成β转角。 1.1.1 蛋白质二级结构预测方法 蛋白质二级结构的预测通常被认为是蛋白结构预测的第一步,不同的氨基酸残基具有不同的二级结构形成倾向性。预测蛋白质二级结构的算法大多以已知三维结构和二级结构的蛋白质为依据,根据蛋白质序列中每个氨基酸的相对保守水平,通过人工神经网络、隐马可夫模型等算法构建预测方法。总的来说,二级结构预测仍是未能完全解决的问题,一般对于α螺旋预测精度较好,对β折叠差些,而对除α螺旋和β折叠等之外的无规则二级结构则效果很差。 目前较为常用的几种方法有:PHD、GOR、Predator等,其中PHD被认为是二级结构预测的标准,它的预测准确率能达到70%。PHD整合了许多神经网络的结果,每个结果都是根据局部序列上下文关系和整体蛋白质性质(蛋白质长度、氨基酸频率等)来预测残基的二级结构。最终的预测是所有这些神经网络输出的算术平均值,这种整合方案被称为陪审团决定(jury decision)或赢家通吃(winner-take-all)法。 蛋白质预测软件的输出结果一般与下面的结果相似:

图 8 4 二级结构预测结果 图 8 4中H代表螺旋(Alpha helix), E代表折叠(Extended strand), C代表无规则卷曲(Random coil)等。这是预测软件GOR (https://npsa-prabi.ibcp.fr/cgi-bin/secpred_gor4.pl) 的结果。
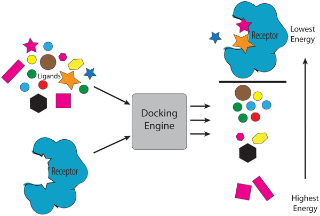
分子对接
- 受体-配体分子对接的一般步骤:
- 受体和配体分子处理
- 寻找潜在的结合口袋
- 旋转平移配体,生成一系列配体结合姿势(pose);对于柔性对接,配体本身的构象变化也要考虑
- 对每个结合姿势的结合能进行估计
- 对结合姿势聚类与排名
References
- Jacob RB, Andersen T, McDougal OM (2012) Accessible High-Throughput Virtual Screening Molecular Docking Software for Students and Educators. PLoS Comput Biol 8(5): e1002499. https://doi.org/10.1371/journal.pcbi.1002499